2012, oxford academic bioinformatics, 86 citation (6.931)
https://academic.oup.com/bioinformatics/article/28/13/1790/234696
CNVRuler: a copy number variation-based case–control association analysis tool
Abstract. Summary: The method for genome-wide association study (GWAS) based on copy number variation (CNV) is not as well established as that for single nucleo
academic.oup.com
Abstract
- CNV기반 GWAS 분석 및 CNV 연구를 위한 여러가지 도구들이 존재한다.
- 하지만 CNV 관련 연구에 필수적인 CNVRs(CNV regions)에 대한 적절한 정의를 제공하지 않는다.
- CNVRuler 를 제안한다.
- 가장 일반적인 CNV 알고리즘 10개의 결과(output file)를 직접적으로 CNVRuler input file로 사용할 수 있다.
- 3가지 다른 CNVR에대한 정의를 사용해 결과를 만들어낼 수 있다.
- 인구 계층화(stratification)를 위한 4가지 통계 연관 테스트 및 옵션을 지원한다.
Introduction
Abstract과 동일
Methods
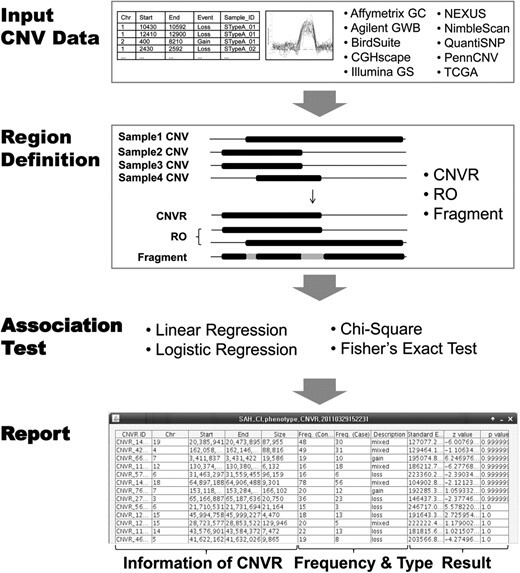
Building CNVRs
공통 CNV 영역의 세 가지 다른 정의를 지원한다.
1. CNVR(병합)
- CNVR 내 총 기여한 CNV의 10% 미만(optional)이 차지하는 영역은 제거
- 개별 CNV는 적어도 1 bp 중첩되는 CNV를 덮는 게놈 영역인 CNVR로 병합된다.
(option1) Remove smaller than
(option2) Recurrence : 임계값을 낮추면 더 많은 희소 영역이 제거되고, 임계값을 높이면 더 적은 희소 영역이 제거됩니다.
2. RO(reciprocal overlap)
- 공통 CNV 영역은 상호 중첩(reciprocal overlap, RO)을 통해도 결정될 수 dlTek.
- 50% 이상 중첩된 CNV는 하나로 통합합니다.
3. 중첩되는 모든 지역을 분할한다.
유효성을 검증 데이터는, 10명의 개인의 Affymetrix SNP array6.0유전자 분석 데이터에서 식별된 CNV, CNVRuler, CONAN을 이용해 정의된 CNVRs이다.
위 세가지 방법으로 정의된 CNVR은 CONAN에 의해 정의된 CNVR과 100일치한다.
Association analysis
정의된 CNVR과 임상 정보를 사용해 5가지 통계적 검정을 지원한다.
- chi-squared and Fisher's exact tests in addition to logistic and linear regression analyses
CNVRuler의 성능을 검증
- Illumina HumanHap300 BeadChip을 사용하여 서발성 동맥류 출혈 500명의 경우에서 4574개의 CNV 데이터를 적용
- 1843 CNVR, 2211 RO 및 2797 fragment 를 식별함
(CNV 분포가 균일하지 않은 경우 Fragment 알고리즘이 더 적절할 수 있다.
Conclusions
장점:
CNVRuler은 세 가지 다른 CNVR 정의 알고리즘을 지원합니다. 이는 사용자가 자신의 데이터에 가장 적합한 알고리즘을 선택할 수 있도록 합니다.
CNVRuler은 간단한 탭 구분 파일 형식으로 인코딩된 임상 정보를 지원합니다. 이를 사용하여 CNVR과 진단 정보를 연결할 수 있습니다.
CNVRuler은 chi-quared, Fisher's exact test, logistic regression, linear regression 등 다양한 통계 분석 방법을 지원합니다. 사용자는 데이터에 가장 적합한 방법을 선택할 수 있습니다.
CNVRuler은 다중 검정을 위해 FDR (False Discovery Rate) 및 Bonferroni 보정을 지원합니다.
CNVRuler은 population stratification을 위해 주성분 분석 (PCA)을 사용합니다. 이를 통해 사용자는 다른 인구 항목들이 CNVR-표현형 연관 분석에 미치는 영향을 고려할 수 있습니다.
(중첩 비율을 수정할 수 없다는 단점이 있으며, 여러 수치로 테스트해볼 수 없다는 점이 아쉽다.
또한 1bp, 50% 이러한 수치가 나의 데이터에 적절하지 않는 값일 수 있기 때문이다.
하지만 리눅스기반 소프트웨어도 제공해주기 때문에, 위의 파라메터값을 코드상으로 수정해 실행해 볼 수 있을 것 같다.
더불어 여러 통계적 테스트를 제공한다고 하지만, clinical information이 없는 우리 연구에서는 활용성이 떨어진다고 볼 수 있겠다. 따라서 본 연구에서 주장하는 인구의 계층화 분석은 연구에 적용해 보기 어려워 보인다.
단, 우리가 갖고 있는 CNV의 크기, 빈도, CNV 샘플간 유사성과 같은 추가 정보들로 PCA 을 적용해볼 수 있을 것 같긴 하다.



