https://www.biorxiv.org/content/10.1101/2022.10.11.511790v2
이제 막 연구되는 분야 중 하나인 single cell에서의 microbiome.
생물학자들은 어떤 결과를 보고 싶어하고, 어떤 어려움을 겪고있는지 살피기 위함이다.
더불어 어떻게 데이터가 만들어지고 어떤 정보가 있는지 공부하고자 한다.
Motivation & Purpose
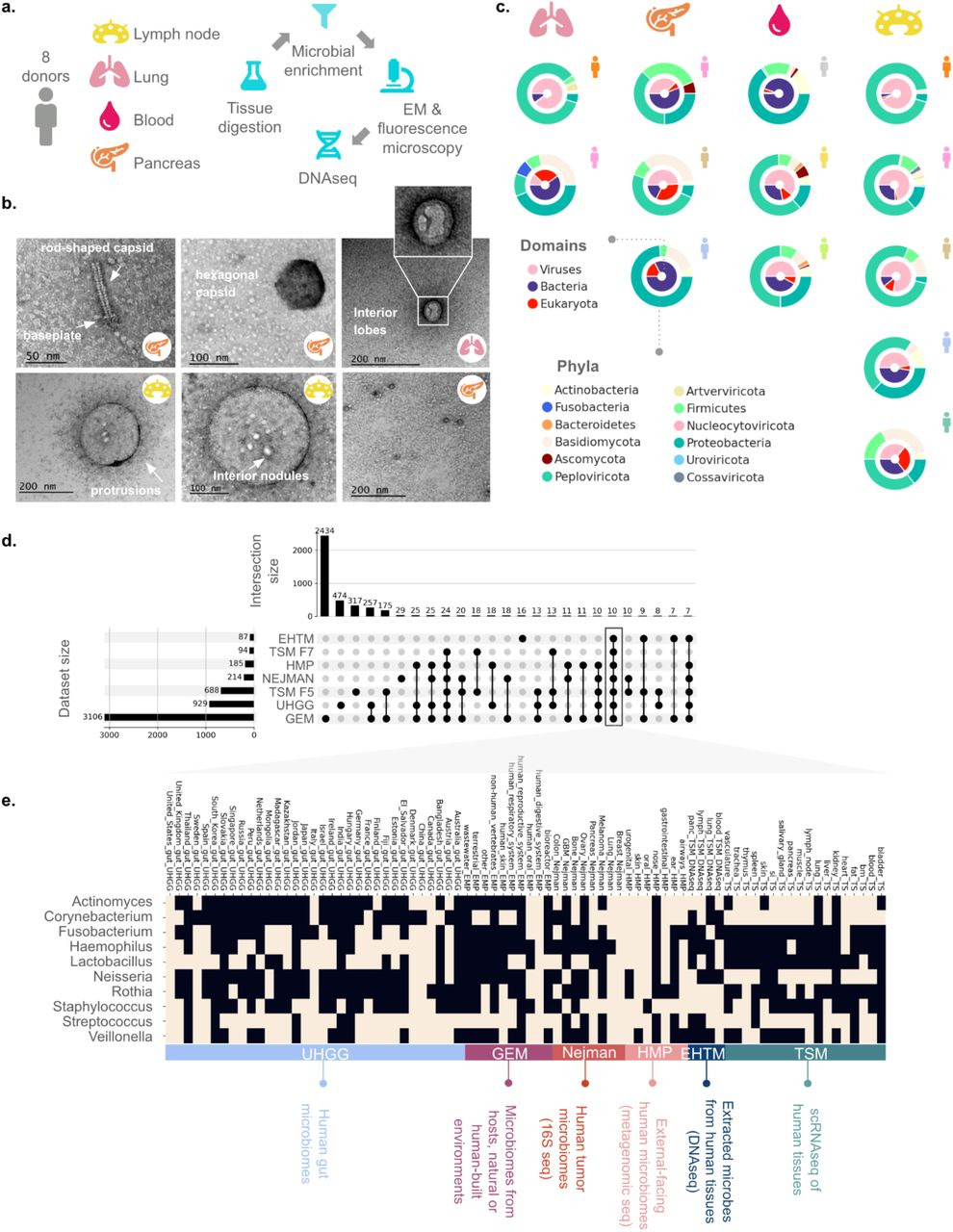
- create an atlas of human tissue microbiome, called Tabula Sapiens Microbiome.
- 11 human organ
- 19 tissue type
- 400,000+ annotaed cells
- 100+ cell types
- ~70 billion sequences from bacterial, viral and fungal species

- 여전히 미지의 기관 및 조직 존재, 그리고 감염성 질병이 아닌 경우의 연구의 부재
- 감염성 질병이 아니면, 미생물이 없다고 추정함
- 의의
- mapping microbial flow routes in body, 예상치 못한 경로의 존재 입증
- tumor/ tumor-free tissues 간의 유의미하게 차이가 있는 bacterial genera 발견
- by increasing the resolution of sampling, cell type 별 host-microbe 네트워크를 밝힘
Novelity
- SIMBA pipeline
- quality filtering
- mapping microbial flow routes
- increase the resolution of tissue microbiomes
- network of human cell types
Materials
- 기존의 Tabulas Sapiens을 viral, baterial and fungal 시퀀스 식별하는 것으로 확장한 것.
- 품질 관리 방법: ML for decontamination and a LLM for habitat classficiaation
- raw single-cell transcriptomic dataset을 이용
- 16개의 외부 데이터와 통합
- for identificaiton of microbioal seuqences
- the Human Gastrointestinal Bacteria Culture Collection (HBC)
- the expanded Human Oral Microbiome Dataset(HOMD)
- Human Microbiome Project (HMP) 등
- for decontamination and analysis
- Narunsky-Haziza et al.(tumor mycobiomes)
- Nejman et al. (tumor microbiomes)
- the Unified Human Gastrointestinal Genome5 (UHGG) collection,
- Genomes from Earth’s Microbiomes (GEM)
- Pathosystems Resource Integration Center(PATRIC)
- for validation
- addtional cohort of 8 donors, called EHTM dataset
Results


Interested in
- 굉장히 단순한 아이디어로 quality control를 사용한 것으로 더 개선하거나 automatics하게 바꿔볼 수 있겠다.
- host genome으로부터 microbiome 데이터를 extract함으로써 더 많은 결과데이터들을 얻을 수 있었던 점.
- 데이터베이스화한 덕분에 추후 해당 분야의 연구(scRNAseq microbiome )가 활발해질 것으로 기대가 되는 점
- 다양한 관점에서 데이터를 분석해, 구체적인 데이터 활용성을 제시하고 기존과 다른 가설을 제안
- 이 것들을 시각화해서 잘 풀어내서 흥미로웠던 논문.
'Paper' 카테고리의 다른 글
| Deep learning in biomedicine (0) | 2025.02.15 |
|---|---|
| EnsembleCNV: an ensemble machine learning algorithm to identify and genotype copy number variation using SNP array data (1) | 2025.01.28 |
| Genomic Language Models: Opportunities and Challenges (0) | 2025.01.19 |
| ECOLE: Learning to call copy number variants on whole exome sequencing data (0) | 2025.01.19 |
| (Archive)Trasnformer 이해하기 (0) | 2024.12.09 |



